megabase
deviation (in bases)
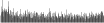
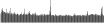
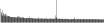
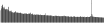
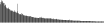
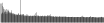

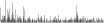
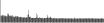
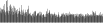
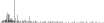
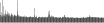
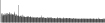
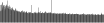
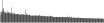

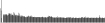
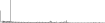
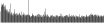
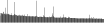
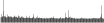
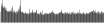
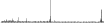
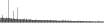
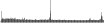
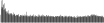
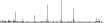
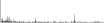
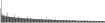
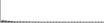


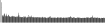
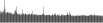
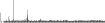
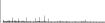
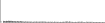
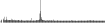

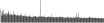
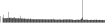

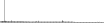

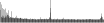
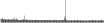
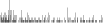
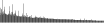
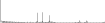
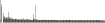
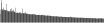
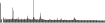
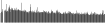
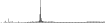
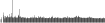
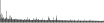
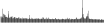
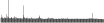
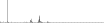
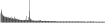
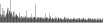
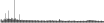
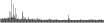
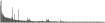
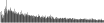
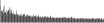
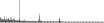
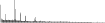
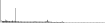
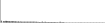
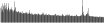
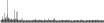
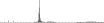
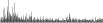
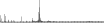
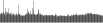
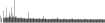
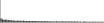
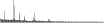
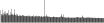
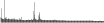
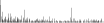
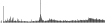


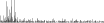

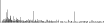
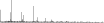
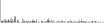
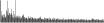
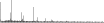
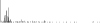
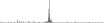
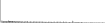
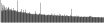
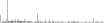
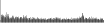
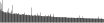
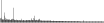

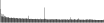
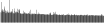
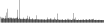
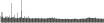
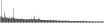
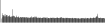
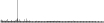
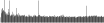
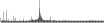
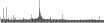
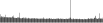

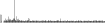

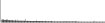
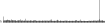
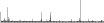
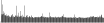
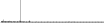
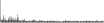
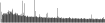
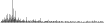
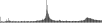
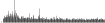
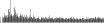
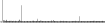
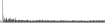
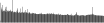
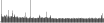

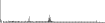
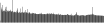
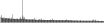
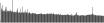
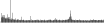
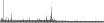
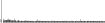
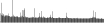
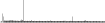
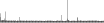


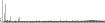
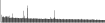

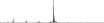

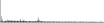
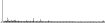
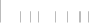
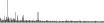
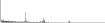

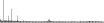
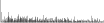
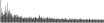

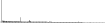
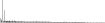


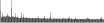
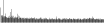
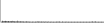

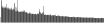
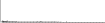

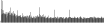
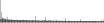
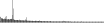
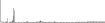


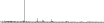

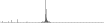

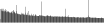
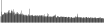
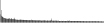
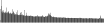

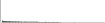
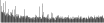
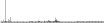
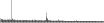
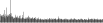
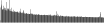
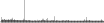
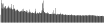
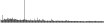
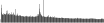
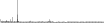
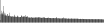
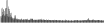
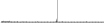
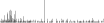
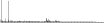
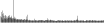
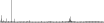
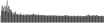
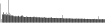
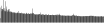
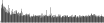
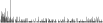
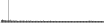
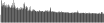
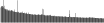
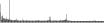
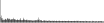
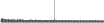
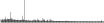
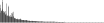

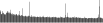
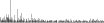
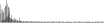
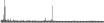
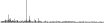

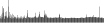
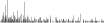
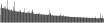
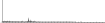
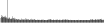
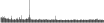
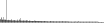
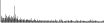
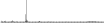
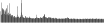
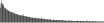

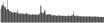
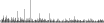
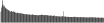
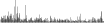
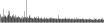
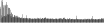
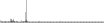
Procedure followed:
- Reference human genome sequence (build37, version 2) is processed.
- Sequence repeat elements are mapped according to NCBI's masking data.
- Gene coordinates and annotations are taken from NCBI's gbk files.
- Unresolved segments are removed, 2.85 billion bases in 289 continuous scaffolds are analyzed.
- Restriction enzyme sites are located, distances between consecutive sites ("fragment lengths") are tabulated.
- Mean and standard deviation of distances, average number of sites per megabase
of sequence are calculated.
- Distribution of site distances plotted for the 1-350 bp range with base resolution (click "details").
- Distribution of distances binnned with standard_deviation/50 resolution plotted (click "details").
- Peaks in these distributons (exceptionally frequent fragment lengths) are identified, and
associated with repeat elements.
- Longest uncut segments are listed for each specificity.
- Repeat content and gene content of longest uncut segments are tabulated.
| Enzyme | Specificity | Sites per megabase |
Distance mean, standard deviation (in bases) |
Distributions | |
|---|---|---|---|---|---|
| AclI | AACGTT | 56.7 | 17647 (18473) | 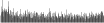 |
details |
| LlaCI | AAGCTT | 292.6 | 3417 (3640) | 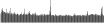 |
details |
| Mae7806I | AAGGAG | 803.3 | 1244 (1373) | 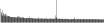 |
details |
| SspI | AATATT | 819.7 | 1219 (1781) | 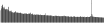 |
details |
| Tsp509I | AATT | 7498.7 | 133 (181) | 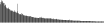 |
details |
| RlaII | ACACAG | 859.5 | 1163 (1251) | 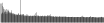 |
details |
| PciI | ACATGT | 369.9 | 2703 (3084) |  |
details |
| AgeI | ACCGGT | 18.4 | 54298 (59099) | 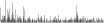 |
details |
| BspMI | ACCTGC | 477.4 | 2094 (2374) | 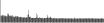 |
details |
| SexAI | ACCWGGT | 103.1 | 9696 (10446) | 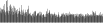 |
details |
| MluI | ACGCGT | 7.3 | 137031 (192274) | 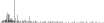 |
details |
| TspGWI | ACGGA | 347.4 | 2878 (3333) | 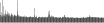 |
details |
| BceSIII | ACGGC | 242.1 | 4129 (5672) | 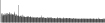 |
details |
| MaeII | ACGT | 751.7 | 1330 (1440) | 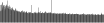 |
details |
| HpyCH4III | ACNGT | 2634.7 | 379 (392) | 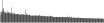 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| BaeI | ACNNNNGTAYC | 91.1 | 10977 (11478) |  |
details |
| BsaXI | ACNNNNNCTCC | 459.0 | 2178 (2468) | 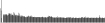 |
details |
| AflIII | ACRYGT | 512.7 | 1950 (2206) | 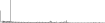 |
details |
| SpeI | ACTAGT | 137.4 | 7277 (8275) | 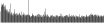 |
details |
| BsrI | ACTGG | 1985.0 | 503 (543) | 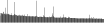 |
details |
| BmrI | ACTGGG | 589.4 | 1696 (1898) | 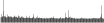 |
details |
| BglII | AGATCT | 269.3 | 3713 (3890) | 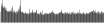 |
details |
| AfeI | AGCGCT | 37.7 | 26506 (34587) | 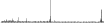 |
details |
| AluI | AGCT | 4477.1 | 223 (236) | 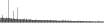 |
details |
| StuI | AGGCCT | 279.7 | 3575 (4249) | 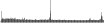 |
details |
| ScaI | AGTACT | 188.5 | 5304 (5918) | 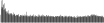 |
details |
| ApyPI | ATCGAC | 33.2 | 30110 (31599) | 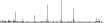 |
details |
| ClaI | ATCGAT | 29.7 | 33626 (35270) | 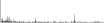 |
details |
| TspDTI | ATGAA | 3622.6 | 276 (337) | 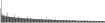 |
details |
| NsiI | ATGCAT | 322.3 | 3102 (3580) | 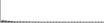 |
details |
| AseI | ATTAAT | 508.6 | 1966 (2711) | 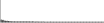 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| SwaI | ATTTAAAT | 78.8 | 12686 (16793) |  |
details |
| OgrI | CAACNAC | 362.7 | 2756 (3162) |  |
details |
| DraRI | CAAGNAC | 436.9 | 2289 (2385) | 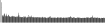 |
details |
| NhaXI | CAAGRAG | 373.2 | 2679 (2824) | 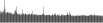 |
details |
| CspCI | CAANNNNNGTGG | 150.1 | 6662 (6799) | 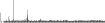 |
details |
| TthHB27I | CAARCA | 1590.7 | 628 (674) | 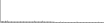 |
details |
| MunI | CAATTG | 191.1 | 5232 (5927) | 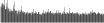 |
details |
| BssSI | CACGAG | 135.5 | 7382 (8789) | 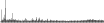 |
details |
| BmgBIB | CACGTC | 94.1 | 10629 (12997) | 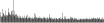 |
details |
| DraIII | CACNNNGTG | 193.5 | 5168 (5767) | 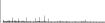 |
details |
| AleI | CACNNNNGTG | 186.7 | 5355 (6183) | 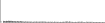 |
details |
| TstI | CACNNNNNNTCC | 570.5 | 1752 (1985) | 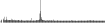 |
details |
| CcoMI | CAGCAG | 815.1 | 1226 (1561) |  |
details |
| SbaI | CAGCTG | 375.4 | 2664 (3230) | 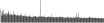 |
details |
| AlwNI | CAGNNNCTG | 486.4 | 2055 (2447) | 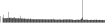 |
details |
| SdeAI | CAGRAG | 2271.7 | 440 (521) |  |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| TspRI | CASTG | 3029.4 | 330 (361) | 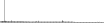 |
details |
| NdeI | CATATG | 313.5 | 3189 (3866) |  |
details |
| NlaCI | CATCAC | 549.4 | 1820 (1944) | 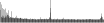 |
details |
| PlaDI | CATCAG | 576.0 | 1736 (1847) | 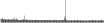 |
details |
| RceI | CATCGAC | 10.2 | 98348 (106383) | 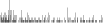 |
details |
| NlaIII | CATG | 4785.6 | 208 (224) | 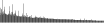 |
details |
| DrdV | CATGNAC | 513.0 | 1949 (2210) | 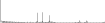 |
details |
| MslI | CAYNNNNRTG | 1130.0 | 884 (949) | 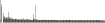 |
details |
| BspNCI | CCAGA | 2499.4 | 400 (446) | 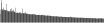 |
details |
| CjeI | CCANNNNNNGT | 1799.1 | 555 (600) | 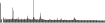 |
details |
| XcmI | CCANNNNNNNNNTGG | 285.3 | 3504 (4010) | 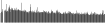 |
details |
| BstXI | CCANNNNNNTGG | 630.8 | 1585 (1952) | 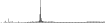 |
details |
| PflMI | CCANNNNNTGG | 340.6 | 2936 (3306) | 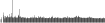 |
details |
| McaCI | CCATC | 2130.2 | 469 (526) | 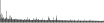 |
details |
| NcoI | CCATGG | 262.6 | 3807 (4230) | 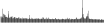 |
details |
| CchIII | CCCAAG | 622.0 | 1607 (1836) | 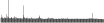 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| BseYIB | CCCAGC | 1282.9 | 779 (1212) | 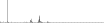 |
details |
| FauI | CCCGC | 493.5 | 2026 (3707) | 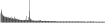 |
details |
| RpaBI | CCCGCAG | 32.3 | 30961 (55857) | 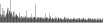 |
details |
| XmaI | CCCGGG | 130.9 | 7637 (12676) | 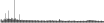 |
details |
| BscGI | CCCGT | 374.6 | 2669 (3657) | 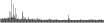 |
details |
| AciI | CCGC | 1436.2 | 696 (1292) | 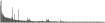 |
details |
| SenpCI | CCGCGG | 22.8 | 43860 (107870) | 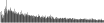 |
details |
| BsrBI | CCGCTC | 79.8 | 12526 (19735) | 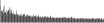 |
details |
| MspI | CCGG | 802.7 | 1245 (2198) | 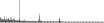 |
details |
| StyD4I | CCNGG | 4102.8 | 243 (390) | 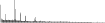 |
details |
| Hpy99IV | CCNNGG | 3564.0 | 280 (421) | 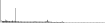 |
details |
| BslIB | CCNNNNNNNGG | 2619.4 | 381 (551) | 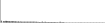 |
details |
| BtgI | CCRYGG | 405.0 | 2469 (3051) | 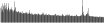 |
details |
| NciI | CCSGG | 677.0 | 1477 (3121) | 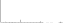 |
details |
| AvrII | CCTAGG | 206.5 | 4842 (5176) | 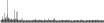 |
details |
| MnlI | CCTC | 9514.9 | 105 (141) | 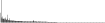 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| BbvCIB | CCTCAGC | 485.7 | 2058 (2797) | 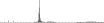 |
details |
| SdaI | CCTGCAGG | 26.9 | 37190 (53243) | 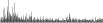 |
details |
| Bpu10IB | CCTNAGC | 834.6 | 1198 (1496) | 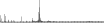 |
details |
| Bsu36I | CCTNAGG | 328.0 | 3048 (3496) | 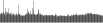 |
details |
| EcoNI | CCTNNNNNAGG | 268.5 | 3723 (4408) | 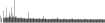 |
details |
| HpyAV | CCTTC | 2403.1 | 416 (499) | 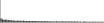 |
details |
| PspGI | CCWGG | 3425.7 | 291 (431) | 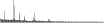 |
details |
| StyI | CCWWGG | 1079.6 | 926 (1055) | 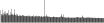 |
details |
| PspPRI | CCYCAG | 1822.8 | 548 (719) | 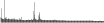 |
details |
| SstE37I | CGAAGAC | 15.1 | 66159 (70539) | 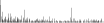 |
details |
| BcgI | CGANNNNNNTGC | 111.9 | 8933 (11966) | 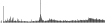 |
details |
| XorKI | CGATCG | 4.2 | 235598 (295546) |  |
details |
| PspOMII | CGCCCAR | 140.4 | 7124 (11163) |  |
details |
| PliMI | CGCCGAC | 4.2 | 238009 (432664) | 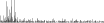 |
details |
| TmaI | CGCG | 253.5 | 3944 (8713) |  |
details |
| GauT27I | CGCGCAGG | 3.0 | 338220 (587467) | 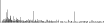 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| EagI | CGGCCG | 31.0 | 32223 (67311) | 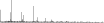 |
details |
| RsrII | CGGWCCG | 3.3 | 304917 (506969) | 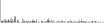 |
details |
| RpaB5I | CGRGGAC | 42.3 | 23637 (37962) | 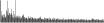 |
details |
| BsiEI | CGRYCG | 47.1 | 21208 (41607) | 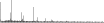 |
details |
| BsiWI | CGTACG | 4.0 | 252613 (300468) | 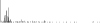 |
details |
| Esp3I | CGTCTC | 224.1 | 4462 (6371) | 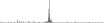 |
details |
| Hpy99I | CGWCG | 53.9 | 18542 (30957) | 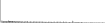 |
details |
| MspA1I | CMGCKG | 502.3 | 1990 (2663) | 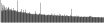 |
details |
| SgrAI | CRCCGGYG | 5.2 | 192930 (297350) | 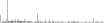 |
details |
| MaqI | CRTTGAC | 96.8 | 10331 (10900) | 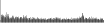 |
details |
| MthZI | CTAG | 2716.1 | 368 (389) | 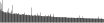 |
details |
| BspCNI | CTCAG | 3282.8 | 304 (357) | 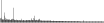 |
details |
| XhoI | CTCGAG | 41.9 | 23844 (28573) |  |
details |
| TdeII | CTCTTC | 789.5 | 1266 (1391) | 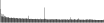 |
details |
| Eco57I | CTGAAG | 749.7 | 1333 (1412) | 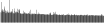 |
details |
| XveI | CTGCAG | 452.8 | 2208 (2696) | 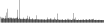 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| BpmI | CTGGAG | 1011.4 | 988 (1140) | 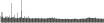 |
details |
| HpyHI | CTNAG | 4843.9 | 206 (222) | 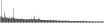 |
details |
| LlaGI | CTNGAYG | 433.6 | 2306 (2395) | 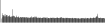 |
details |
| SfeI | CTRYAG | 1209.3 | 826 (864) | 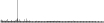 |
details |
| AflII | CTTAAG | 223.9 | 4466 (4755) | 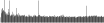 |
details |
| BpuEI | CTTGAG | 678.0 | 1474 (1599) | 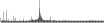 |
details |
| SmlI | CTYRAG | 944.0 | 1059 (1112) | 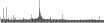 |
details |
| RpaB53I | CWTCCAG | 381.6 | 2620 (2846) | 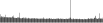 |
details |
| NspIII | CYCGRG | 349.1 | 2864 (4569) |  |
details |
| PpiI | GAACNNNNNCTC | 86.3 | 11582 (12233) | 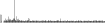 |
details |
| AloI | GAACNNNNNNTCC | 85.0 | 11771 (12424) |  |
details |
| NcuI | GAAGA | 3071.0 | 325 (371) | 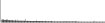 |
details |
| XmnI | GAANNNNTTC | 346.6 | 2884 (3093) | 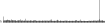 |
details |
| Mva1269I | GAATGC | 428.7 | 2332 (2479) | 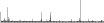 |
details |
| Van91II | GAATTC | 272.1 | 3674 (3884) | 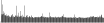 |
details |
| EsaSSI | GACCAC | 309.3 | 3232 (3570) | 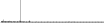 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| TaqII | GACCGA | 44.9 | 22253 (26740) | 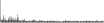 |
details |
| NmeBI | GACGC | 195.9 | 5104 (7510) | 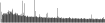 |
details |
| ZraI | GACGTC | 24.4 | 41041 (50398) | 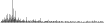 |
details |
| Tth111I | GACNNNGTC | 245.7 | 4069 (5193) | 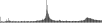 |
details |
| PshAI | GACNNNNGTC | 93.6 | 10678 (11530) | 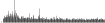 |
details |
| AhdI | GACNNNNNGTC | 94.9 | 10542 (11087) | 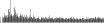 |
details |
| NgoAVIII | GACNNNNNTGA | 431.0 | 2319 (2390) | 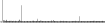 |
details |
| SdeOSI | GACNNNNRTGA | 197.0 | 5075 (5131) | 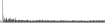 |
details |
| R2.BspD6I | GACTC | 1359.0 | 735 (799) | 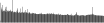 |
details |
| SacI | GAGCTC | 208.0 | 4807 (5456) | 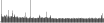 |
details |
| MchCM4I | GAGGAG | 750.4 | 1332 (1797) |  |
details |
| CjeNII | GAGNNNNNGT | 2187.8 | 457 (541) | 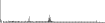 |
details |
| PleI | GAGTC | 1359.0 | 735 (799) | 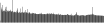 |
details |
| HpyAIV | GANTC | 2999.2 | 333 (343) | 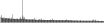 |
details |
| - | GASTC | 1359.0 | 735 (799) | 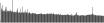 |
details |
| EcoRV | GATATC | 150.5 | 6644 (7674) | 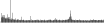 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| Sth368I | GATC | 2490.9 | 401 (419) | 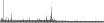 |
details |
| MamI | GATNNNNATC | 155.4 | 6436 (6799) | 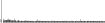 |
details |
| TfiI | GAWTC | 1640.1 | 609 (635) | 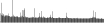 |
details |
| HaeIV | GAYNNNNNRTC | 476.6 | 2098 (2175) | 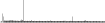 |
details |
| R2.BsrDI | GCAATG | 494.1 | 2023 (2077) | 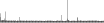 |
details |
| Lsp1109I | GCAGC | 1816.9 | 550 (745) |  |
details |
| BtsIB | GCAGTG | 1028.7 | 972 (1246) |  |
details |
| BstAPI | GCANNNNNTGC | 284.8 | 3511 (3769) | 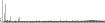 |
details |
| SfaNI | GCATC | 1258.3 | 794 (870) | 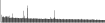 |
details |
| SphI | GCATGC | 190.7 | 5244 (5663) |  |
details |
| NmeAIII | GCCGAG | 272.6 | 3668 (5946) | 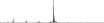 |
details |
| AspCNI | GCCGC | 313.6 | 3188 (6458) |  |
details |
| NgoMIV | GCCGGC | 43.4 | 23039 (44459) | 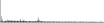 |
details |
| RspRSI | GCCNNNNNGGC | 183.2 | 5459 (7941) | 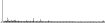 |
details |
| AsiSI | GCGATCGC | 0.4 | 2676658 (2805325) | 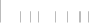 |
details |
| BtgZI | GCGATG | 62.2 | 16074 (19118) | 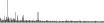 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| Hpy99III | GCGC | 578.6 | 1728 (3147) | 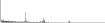 |
details |
| McaTI | GCGCGC | 25.1 | 39882 (94447) |  |
details |
| CdpI | GCGGAG | 161.4 | 6197 (9382) | 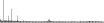 |
details |
| NotI | GCGGCCGC | 3.3 | 302116 (600864) | 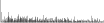 |
details |
| SpoDI | GCGGRAG | 70.6 | 14154 (21809) | 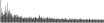 |
details |
| LlaDII | GCNGC | 2130.6 | 469 (681) |  |
details |
| Cac8I | GCNNGC | 1887.9 | 529 (719) | 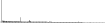 |
details |
| MwoI | GCNNNNNNNGC | 2131.2 | 469 (642) | 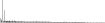 |
details |
| NgoFVII | GCSGC | 313.6 | 3188 (6458) |  |
details |
| NheI | GCTAGC | 98.2 | 10180 (11291) |  |
details |
| AmaCSI | GCTCCA | 471.2 | 2122 (2546) | 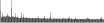 |
details |
| SapI | GCTCTTC | 130.5 | 7664 (8282) | 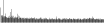 |
details |
| BlpI | GCTNAGC | 183.2 | 5459 (6564) | 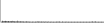 |
details |
| TseI | GCWGC | 1816.9 | 550 (745) |  |
details |
| Bsp1286I | GDGCHC | 1678.4 | 595 (780) | 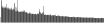 |
details |
| CchII | GGARGA | 1724.9 | 579 (687) | 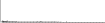 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| EacI | GGATC | 1171.4 | 853 (991) |  |
details |
| OkrAI | GGATCC | 126.1 | 7927 (8692) | 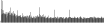 |
details |
| StsI | GGATG | 1792.0 | 558 (625) | 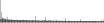 |
details |
| SuaI | GGCC | 2918.3 | 342 (478) | 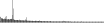 |
details |
| HauI | GGCCAAG | 208.8 | 4788 (6098) | 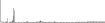 |
details |
| FseI | GGCCGGCC | 4.6 | 215446 (390072) |  |
details |
| SfiI | GGCCNNNNNGGCC | 16.3 | 61440 (99839) |  |
details |
| SfoI | GGCGCC | 80.0 | 12499 (19651) | 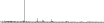 |
details |
| AscI | GGCGCGCC | 1.6 | 640695 (1072205) |  |
details |
| EciI | GGCGGA | 198.1 | 5047 (8085) | 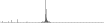 |
details |
| BloAORF92P | GGCGGC | 121.5 | 8231 (18398) |  |
details |
| BspLU11III | GGGAC | 1328.7 | 752 (925) | 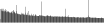 |
details |
| PspOMI | GGGCCC | 159.8 | 6257 (10138) | 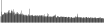 |
details |
| AteTI | GGGRAG | 1416.8 | 705 (958) | 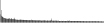 |
details |
| R2.BssIMI | GGGTC | 1094.0 | 914 (1168) | 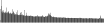 |
details |
| TdeIII | GGNCC | 2198.6 | 454 (700) |  |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| NlaIV | GGNNCC | 2466.6 | 405 (604) | 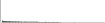 |
details |
| KpnI | GGTACC | 100.1 | 9992 (10869) | 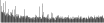 |
details |
| Eco31I | GGTCTC | 576.0 | 1736 (2130) | 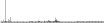 |
details |
| NgoBVIII | GGTGA | 2060.4 | 485 (530) | 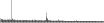 |
details |
| BstEII | GGTNACC | 116.7 | 8568 (9416) | 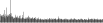 |
details |
| SinI | GGWCC | 932.9 | 1071 (1381) | 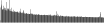 |
details |
| HgiCI | GGYRCC | 539.1 | 1854 (2280) | 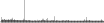 |
details |
| Bme1580I | GKGCMC | 692.2 | 1444 (1899) | 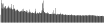 |
details |
| HgiGI | GRCGYC | 159.9 | 6254 (9605) | 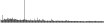 |
details |
| EcoT38I | GRGCYC | 814.4 | 1227 (1701) | 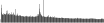 |
details |
| RpaTI | GRTGGAG | 465.5 | 2148 (2589) | 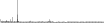 |
details |
| RsaI | GTAC | 1763.8 | 566 (606) | 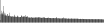 |
details |
| BstZ17I | GTATAC | 148.3 | 6743 (8247) | 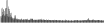 |
details |
| BciVI | GTATCC | 239.4 | 4177 (4407) | 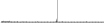 |
details |
| XamI | GTCGAC | 10.7 | 93276 (101295) | 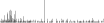 |
details |
| BsmAI | GTCTC | 2295.8 | 435 (534) | 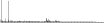 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| ApaLI | GTGCAC | 171.4 | 5833 (6348) | 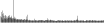 |
details |
| BsgI | GTGCAG | 712.4 | 1403 (1600) | 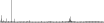 |
details |
| AccI | GTMKAC | 376.3 | 2657 (2820) | 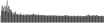 |
details |
| MjaIV | GTNNAC | 2166.8 | 461 (478) | 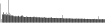 |
details |
| Tsp45I | GTSAC | 1376.8 | 726 (804) | 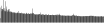 |
details |
| HpaI | GTTAAC | 135.3 | 7389 (8047) | 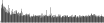 |
details |
| PmeI | GTTTAAAC | 14.3 | 69773 (75434) | 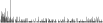 |
details |
| HindII | GTYRAC | 383.4 | 2608 (2748) | 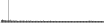 |
details |
| BsiHKAI | GWGCWC | 711.1 | 1406 (1600) | 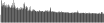 |
details |
| ApoI | RAATTY | 2174.0 | 459 (592) | 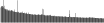 |
details |
| NspI | RCATGY | 1074.5 | 930 (1020) | 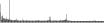 |
details |
| NmeDI | RCCGGY | 113.8 | 8789 (13646) | 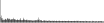 |
details |
| XhoII | RGATCY | 748.2 | 1336 (1393) | 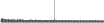 |
details |
| NgoBI | RGCGCY | 192.0 | 5209 (8358) | 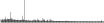 |
details |
| CviKI-1 | RGCY | 15480.8 | 64 (81) | 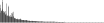 |
details |
| EcoO109I | RGGNCCY | 811.1 | 1232 (1879) |  |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| PpuMI | RGGWCCY | 376.9 | 2653 (3462) | 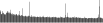 |
details |
| DrdIV | TACGAC | 30.1 | 33258 (34457) | 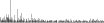 |
details |
| SnaBI | TACGTA | 44.5 | 22493 (25247) | 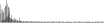 |
details |
| BspHI | TCATGA | 338.5 | 2954 (3161) | 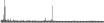 |
details |
| Kpn2I | TCCGGA | 33.0 | 30266 (37714) | 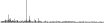 |
details |
| MmeI | TCCRAC | 444.8 | 2248 (2333) |  |
details |
| TthHB8I | TCGA | 528.6 | 1891 (2087) | 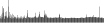 |
details |
| Sbo13I | TCGCGA | 5.0 | 198470 (255722) | 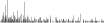 |
details |
| HpyF17I | TCNGA | 2981.6 | 335 (362) | 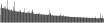 |
details |
| HpyNSH57II | TCNNGA | 3552.0 | 281 (298) | 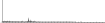 |
details |
| XbaI | TCTAGA | 279.3 | 3580 (3831) | 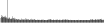 |
details |
| BclI | TGATCA | 255.5 | 3913 (4075) | 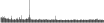 |
details |
| HpyCH4V | TGCA | 4993.6 | 200 (209) | 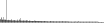 |
details |
| FspI | TGCGCA | 30.6 | 32668 (40144) | 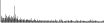 |
details |
| MscI | TGGCCA | 453.2 | 2206 (2513) | 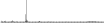 |
details |
| BsrGI | TGTACA | 288.2 | 3469 (3899) | 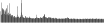 |
details |
| Enzyme | Specificity | Sites per Mbp | Distance mean, std | Distributions | |
| MseI | TTAA | 6656.2 | 150 (214) | 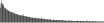 |
details |
| PacI | TTAATTAA | 55.4 | 18038 (22316) |  |
details |
| PsiI | TTATAA | 572.2 | 1747 (2462) | 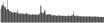 |
details |
| NspV | TTCGAA | 36.7 | 27232 (28088) | 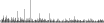 |
details |
| DraI | TTTAAA | 1189.6 | 840 (1133) | 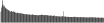 |
details |
| PspXI | VCTCGAGB | 20.4 | 49047 (62813) | 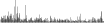 |
details |
| BsaWI | WCCGGW | 96.5 | 10362 (12538) | 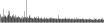 |
details |
| BsaAI | YACGTR | 242.0 | 4131 (4481) | 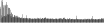 |
details |
| EcoHK31I | YGGCCR | 603.0 | 1658 (2054) | 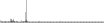 |
details |